Sinergia ed ortogonalità per la caratterizzazione delle proteine
Le macromolecole e gli assemblaggi macromolecolari (generalmente quando > 5000 Dalton), come le proteine, gli acidi nucleici e i lipidi, possono formare strutture ben definite che subiscono cambiamenti conformazionali che inducono riarrangiamenti strutturali influenzati da innumerevoli fattori, intrinseci o estrinseci che ne possono influenzare l’attività. Nel dinamico campo della ricerca biofarmaceutica, la struttura e quindi la stabilità delle proteine sono fattori critici che influenza l’efficacia, la sicurezza e la conservabilità dei prodotti. Per valutarli, due tecniche si distinguono per la loro rilevanza: la Fluorimetria a Scansione Differenziale (DSF) e la Calorimetria a Scansione Differenziale (DSC). Sebbene entrambe forniscano preziose informazioni sulla stabilità termica delle proteine, presentano metodologie, vantaggi e limitazioni distinti che le rendono ortogonali e complementari.
Fluorimetria a Scansione Differenziale (DSF)
La DSF è una tecnica che monitora la denaturazione delle proteine rilevando i cambiamenti nella fluorescenza intrinseca o tramite l’impiego di appositi coloranti.
Con l’aumento della temperatura, la struttura delle proteine va in contro ad un processo di dispiegamento che porta alla variazione dell’esposizione di amminoacidi intrinsecamente fluorescenti, come il triptofano (Fig. 1), generando un segnale che permette di monitorare il processo di unfolding (Fig. 2). In assenza di sufficiente fluorescenza intrinseca è possibile impiegare appositi coloranti fluorescenti che si legano alle regioni idrofobiche esposte della proteina in fase di denaturazione.
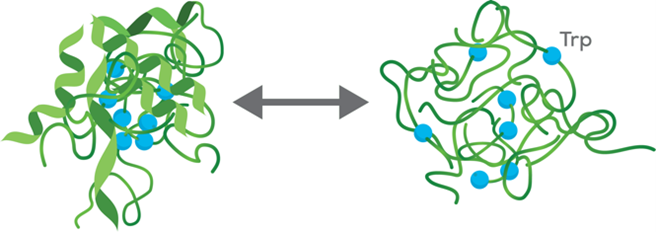
Figura 1 Per le proteine, lo spettro della fluorescenza emessa dipende dall’ambiente locale intorno agli aminoacidi fluorescenti. Lo spettro di fluorescenza di una proteina ripiegata con tasche idrofobiche altamente non polari avrà un aspetto diverso dallo spettro di una proteina non ripiegata con gli aminoacidi fluorescenti esposti a un ambiente acquoso più polare
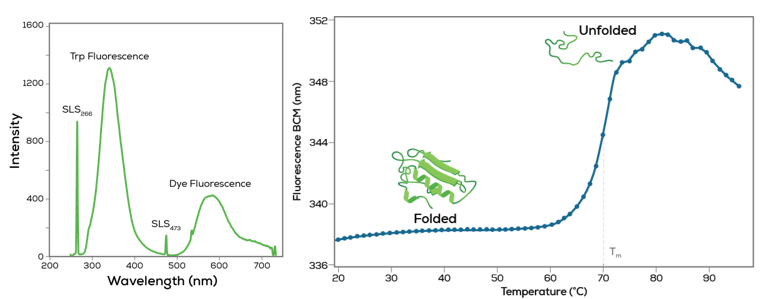
Figura 2 La tecnica DSF funziona misurando le variazioni di fluorescenza al variare della conformazione di una proteina. La fluorescenza può essere quella degli aminoacidi presenti nella proteina (principalmente triptofano, questa è la fluorescenza intrinseca) o di un fluoroforo reporter aggiunto, come SYPRO® Orange. Lo spettro di emissione della proteina cambia quando si scioglie durante l’esposizione al calore. Con la DSF, si misurano le variazioni di fluorescenza quando la proteina si scioglie in risposta all’aumento di temperatura di una rampa termica
Pregi della DSF:
- Elevata produttività (High-throughput): La DSF eccelle negli studi preliminari dove è necessaria una rapida valutazione di numerosi campioni.
- Basso volume di campione: Richiede quantità minime di campione, rendendola ideale per le fasi iniziali della ricerca dove la disponibilità di materiale può essere limitata.
- Velocità: La sua rapidità la rende una scelta pratica per lo screening iniziale.
- Gli strumenti DSF si prestano ad essere combinati con altre tecniche ottiche come la DLS e la SLS consentendono quindi di affiancare informazioni legate caratterizzazione colloidale e allo stato di aggregazione
Difetti della DSF:
- Dipendenza da fluorofori: È limitata alle proteine con fluorofori intrinseci o a quelle che possono essere marcate con un colorante.
- Potenziali interferenze: La presenza di co-soluti, incluso il colorante aggiunto, può interferire con il segnale di fluorescenza, riducendo l’affidabilità dei dati.
- Informazioni limitate: Rileva solo gli eventi di unfolding e non fornisce dati su altri parametri termodinamici essenziali nella comprensione dei meccanismi di stabilità conformazionale, come i cambiamenti entalpici.
Lo strumento Aunty di Unchained Labs (Fig. 3) combina misure di DSF, light scattering statico e light scattering dinamico di campioni in speciali piastre da 96 pozzetti per fornire risultati su denaturazione e aggregazione delle proteine, simultaneamente sullo stesso campione. Se si aggiunge l’analisi personalizzabile attraverso il rilevamento della fluorescenza a spettro completo, si ottiene uno strumento flessibile pronto a rispondere a qualsiasi domanda sulla stabilità delle proteine con una velocità e una qualità dei dati senza pari.

Figura 3 Strumento Aunty e piastre di lettura con formato standard da 96 pozzetti per una manualità più pratica ed un’integrazione completa in flussi di lavoro automatizzati
Calorimetria a Scansione Differenziale (DSC)
La DSC è una tecnica calorimetrica diretta e assoluta che misura il calore assorbito o rilasciato durante il processo di unfolding termico di una proteina, senza la necessità di coloranti fluorescenti. Fornisce un profilo termodinamico completo che include la temperatura di unfolding (Tm), i cambiamenti entalpici (ΔH) e la capacità termica (Cp). La DSC, grazie alla sua “natura” termodicanica può chiarire i fattori che contribuiscono al ripiegamento e alla stabilità delle biomolecole native tre cui le forze deboli, le interazioni idrofobiche, i legami a idrogeno, l’entropia conformazionale e l’ambiente fisico (Fig. 4).

Figura 4 Una biomolecola in soluzione è in equilibrio tra le sue conformazioni native (ripiegate) e denaturate (dispiegate). Più alto è il punto medio di transizione termica (Tm), più stabile è la molecola. La DSC misura l’entalpia (∆H) di dispiegamento che risulta dalla denaturazione indotta dal calore. La DSC può essere impiegata anche per determinare la variazione della capacità termica (ΔCp) della denaturazione.
L’entalpia di unfolding delle proteine è l’area sotto il picco DSC normalizzato per concentrazione e ha unità di misura in calorie (o joule) per mole. In alcuni casi, i modelli termodinamici possono essere adattati ai dati per ottenere l’energia libera di Gibb (ΔG), l’entalpia calorimetrica (ΔHcal), l’entalpia di van’t Hoff (ΔHvH), l’entropia (ΔS) e la variazione della capacità termica (ΔCp) associata alla transizione.
Pregi della DSC:
- Versatilità: Applicabile a un’ampia gamma di biomolecole, inclusi proteine, acidi nucleici, lipidi e loro complessi macromolecolari.
- Misurazione diretta: Non essendo basata su marctori o coloranti, è esente da artefatti causati da quenching, aggregazione proteica o scattering della luce.
- Dati completi e riproducibili: Fornisce dati termodinamici dettagliati e altamente riproducibili, fondamentali per lo sviluppo di formulazioni biologiche, la valutazione di biosimilari e le sottomissioni normative. È spesso considerata il “gold standard” per l’analisi dettagliata della stabilità.
Difetti della DSC:
- Bassa produttività: Non è una tecnica ad alta produttività, rendendola meno adatta per lo screening iniziale di un gran numero di campioni.
- Maggiore volume di campione: Richiede quantitativi di campione (in termini di volumi e massa) più grandi rispetto alla DSF.
Il sistema MicroCal PEAQ-DSC di Malvern Panalytical, nelle sue versioni manuali o automatiche, è il “gold standard” per la caratterizzazione della stabilità di proteine e biomolecole terapeutiche. Con la più alta sensibilità sul mercato, consente l’analisi a partire da soli 10 µg/mL di campione, richiedendone solamente 250 uL (320 µL nel sistema automatizzato). Il software implementa protocolli di analisi automatizzati ed oggettivi che riducono l’intervento del ricercatore nella valutazione dei termogrammi riducendo quindi la variabilità nel processamento dei risultati. Inoltre, i sistemi MicroCal PEAQ-DSC consentono il raggiungimento delle più alte velocità di scansioni presenti sul mercato, fino a 240 °C/h, con notevoli vantaggi in svariati aspetti applicativi come l’aumento della sensibilità e un migliore studio dei comportamenti cinetici.
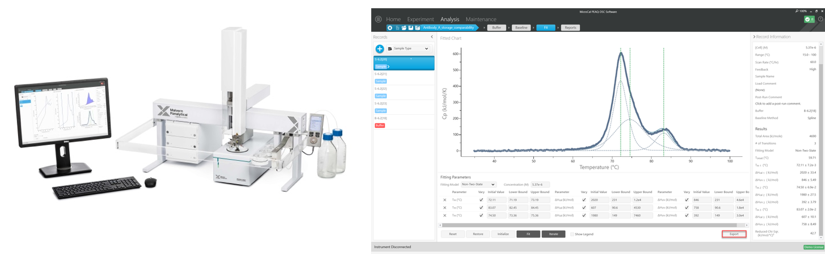
Figura 5 Strumento MicroCal PEAQ-DSC di Malvern Panalytical e schermata rappresentativa del software con selezione delle transizioni e modelli di deconvoluzione.
L’Ortogonalità tra DSC e DSF
L’ortogonalità tra DSC e DSF risiede nella loro capacità di fornire informazioni complementari sulla stabilità delle proteine, utilizzando principi di misurazione fondamentalmente diversi. Sebbene possano sembrare tecniche in competizione è cruciale comprendere come invece dovrebbero essere utilizzate in sinergia per garantire una caratterizzazione più robusta ed una comprensione più approfondita del sistema macromolecolare oggetto di studio.
- Diversi meccanismi di rilevamento: La DSF si basa sui cambiamenti nella fluorescenza indotti dall’esposizione di regioni idrofobiche durante la denaturazione, mentre la DSC misura direttamente i cambiamenti energetici associati a tali transizioni. Questa differenza nei meccanismi di rilevamento riduce la possibilità di artefatti specifici della tecnica.
- Validazione incrociata: I dati ottenuti da entrambe le tecniche possono essere utilizzati per convalidarsi a vicenda. Ad esempio, se entrambe le tecniche indicano un cambiamento simile nella stabilità termica in risposta a una modifica della formulazione, ciò rafforza la fiducia nei risultati.
- Applicazioni strategiche: La DSF può essere impiegata per uno screening rapido e ad alta produttività nelle fasi iniziali dello sviluppo, identificando i candidati più promettenti. Successivamente, la DSC può essere utilizzata per una caratterizzazione termodinamica approfondita dei candidati selezionati, fornendo dati dettagliati per l’ottimizzazione della formulazione e le sottomissioni normative.
Sia la DSF che la DSC sono strumenti indispensabili nella ricerca di base e biofarmaceutica e nella caratterizzazione biofisica della struttura di proteine e complessi macromolecolari. La DSF offre velocità ed efficienza per lo screening ad alta produttività, mentre la DSC fornisce una comprensione termodinamica completa e precisa, essenziale per le fasi avanzate dello sviluppo. Utilizzate in modo strategico, queste tecniche possono fornire una visione ortogonale e completa della stabilità delle proteine, contribuendo in modo significativo allo sviluppo di farmaci biologici sicuri ed efficaci.

